了解与研究蛋白质间的相互作用(Protein-Protein Interaction, PPI),是理解细胞内发生若干生物化学活动的第一步。在众多的PPI研究方法中,通常将蛋白质分为诱饵蛋白与猎物蛋白,利用酵母的生长(Y2H),观察活体细胞中的荧光(荧光能量转移,Fluorescence Resonance Energy Transfer,FRET或双分子荧光互补,Bimolecular Fluorescent Complementation,BiFC)以及采用各类体内与体外的沉淀法(免疫沉淀或亲和纯化等)等方法,检测诱饵蛋白与猎物蛋白之间的相互作用【1】。
难以避免的是,每种方法都具有局限性,必须基于一种以上的PPI方法,才可以得出可信的结果【1】。虽然,这些方法都在植物PPI的研究中较为广泛的应用,但是,我们仍需要发展新的PPI方法,尤其能够解决现有方法的局限性。雷帕霉素(Rapamycin)作为一种化学分子,能够介导分别含有FKBP(FK506-Binding Protein)结构域与FRB(FKBP-Rapamycin-Binding)结构域的蛋白形成异二聚体。雷帕霉素与FKBP结构域结合形成复合体,并进一步与FRB结构域结合。
利用这一特性,前人在人类细胞中创建了用于研究蛋白功能的Knocksideways系统【2】。将线粒体定位信号、荧光蛋白与FRB结合为线粒体锚定蛋白(图1,绿色蛋白),同时,将待检测蛋白与FKBP结构域相连(图1,红色蛋白),在施加雷帕霉素后,待检测蛋白会重定位于线粒体中。由于待检测蛋白的非正常定位,无法发挥其功能,配合其他检测方法可以研究其功能【2】,可用于研究对象突变体致死的环境下。雷帕霉素能够形成二聚体的作用已经在不同类型的生物体中得以应用【3-5】,但少见于植物细胞中【1】。
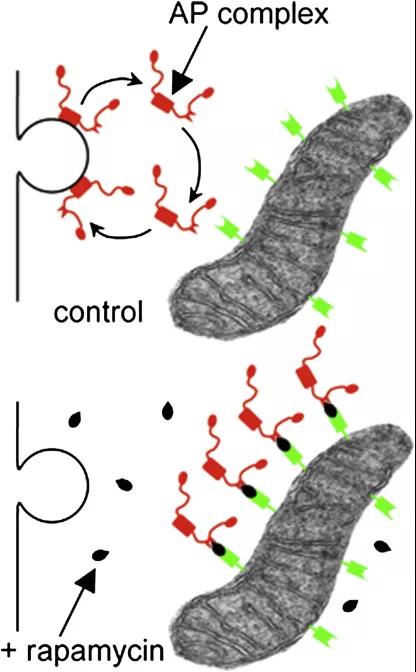
图1 Knocksideways系统【2】
|
|
2021年1月25日,来自比利时根特大学Daniël Van Damme团队在 The Plant Cell发表了题为Visualizing protein-protein interactions in plants by rapamycin-dependent delocalization的研究论文。该研究利用雷帕霉素能够调控异二聚体在细胞内的形成这一过程,创建若干不同细胞结构定位信号的FRB融合蛋白,利用荧光共聚焦显微镜,选取已知互作蛋白,验证并建立,可应用于植物细胞中的KSP(Knocksideways in plants)蛋白互作检测系统,并且探讨了其适用性与局限性,提出了进一步改进的方法。
在该研究中,作者基于Knocksideways系统【2】,结合已知的研究结果,利用MultiSite Gateway技术,创建了融合不同细胞结构定位信号的FRB蛋白(定位信号-荧光分子-FRB),包括微管、细胞核、线粒体以及细胞膜。在荧光共聚焦显微镜的观察下,烟草表皮细胞中,连接不同信号分子的FRB融合蛋白都能够定位于相应的细胞结构上。作者进一步,将细胞结构定位与FRB融合蛋白不同的诱饵蛋白,连接 FKBP蛋白(诱饵蛋白-mCherry-FKBP或FKBP-mCherry-诱饵蛋白),共同在烟草中表达两个融合蛋白。施用雷帕霉素后,融合FKBP的诱饵蛋白在雷帕霉素的作用下,重新定位于含有FRB融合蛋白的细胞结构上。至此,验证了雷帕霉素在植物细胞中,能够介导异二聚体形成。在此基础上,作者利用已知的蛋白相互作用,将猎物蛋白与GFP蛋白相连,在施加雷帕霉素后,猎物蛋白与诱饵蛋白能够共定位在特定细胞结构上。比如,利用KSP系统,重现了SACL蛋白与LHW蛋白的互作【6】。
在雷帕霉素处理后,荧光信号位于细胞核中的SACL-GFP与LHW-mCherry-FKBP,转移到了线粒体上,与线粒体定位的MITO-TagBFP2-FRB信号重叠。基于以上结果,表明KSP系统具有可诱导性。此外,为了探讨KSP的可逆性与稳定性,作者引入了雷帕霉素的拮抗物子囊霉素(Ascomycin),其能够竞争性结合FKBP结构域【7】。实验结果表明,在雷帕霉素处理后,5分钟内就可以观察到荧光信号的重定位现象。在此基础上,再经历子囊霉素处理,荧光信号重定位现象仍然能在24小时后观察到。为了对蛋白质重定位进行定量分析,作者也提供了基于ImageJ/Fiji软件的脚本与操作手册,在图片标准化处理后,配合脚本实现了图片批量处理,可以对施加雷帕霉素前后,荧光信号位置变化进行相对定量。随后,作者也探讨了KSP的局限性。在该系统中,定位于细胞核中的KRP2蛋白,在雷帕霉素处理后,无法观察到较为明显的重定位现象。
最后,作者探讨了KSP系统的前景与改进方向。比如两个以上蛋白间的相互作用,特定细胞区域中酶促反应研究与条件性“敲除”基因等。在改进方向上,提出通过引入组织特异性启动子,或将多个表达框整合到同一载体上等方法,有望拓展KSP系统的适用性。
总而言之,基于文章中实验结果,KSP系统是一个在植物细胞中研究蛋白质相互作用的有力工具。作者通过改进Knocksideways系统【2】,使之较为系统性地应用于植物细胞中PPI研究,相比较于其他适用于植物细胞中的系统具有特定优势。1. 雷帕霉素施用与否,本身可以作为实验对照。在施用后,极短时间内就能观测到结果,且可持续至24小时。2. KSP相较于BiFC与FRET,其相互作用结果读取,并不依赖荧光基团的互补或基团之间的空间距离。但是,KSP系统在检测蛋白互作中具有一定的局限性。相对于可移动性较高的蛋白(如定位于微管与细胞质),或具有多种亚细胞定位的蛋白,有特定且稳定分布的蛋白(比如位于细胞核的KRP2), KSP的作用不明显。再者,KSP表现出可以检测两个以上蛋白的相互作用,但无法了解第三个蛋白是促进或竞争结合。有需要的读者,可以参考近期发表在Plant Physiology上利用酵母,检测第三个蛋白如何调控蛋白互作的Tri-SUS系统【8】。
参考文献
1. Struk, S., Jacobs, A., Sánchez Martín-Fontecha, E., Gevaert, K., Cubas, P., & Goormachtig, S. (2019). Exploring the protein-protein interaction landscape in plants. Plant, cell & environment, 42(2), 387–409.
2. Robinson, M. S., Sahlender, D. A., & Foster, S. D. (2010). Rapid inactivation of proteins by rapamycin-induced rerouting to mitochondria. Developmental cell, 18(2), 324–331.
3. Putyrski, M., & Schultz, C. (2012). Protein translocation as a tool: The current rapamycin story. FEBS letters, 586(15), 2097–2105.
4. Hughes, K. R., & Waters, A. P. (2017). Rapid inducible protein displacement in Plasmodiumin vivo and in vitro using knocksideways technology. Wellcome open research, 2, 18.
5. Wood, L. A., Larocque, G., Clarke, N. I., Sarkar, S., & Royle, S. J. (2017). New tools for "hot-wiring" clathrin-mediated endocytosis with temporal and spatial precision. The Journal of cell biology, 216(12), 4351–4365.
6. Vera-Sirera, F., De Rybel, B., Úrbez, C., Kouklas, E., Pesquera, M., Álvarez-Mahecha, J. C., Minguet, E. G., Tuominen, H., Carbonell, J., Borst, J. W., Weijers, D., & Blázquez, M. A. (2015). A bHLH-Based Feedback Loop Restricts Vascular Cell Proliferation in Plants. Developmental cell, 35(4), 432–443.
7. Li, J. F., Bush, J., Xiong, Y., Li, L., & McCormack, M. (2011). Large-scale protein-protein interaction analysis in Arabidopsis mesophyll protoplasts by split firefly luciferase complementation. PloS one, 6(11), e27364.
8. Zhang, B., Xia, L., Zhang, Y., Wang, H., & Karnik, R. (2020). Tri-SUS: A Yeast Split-Ubiquitin assay to examine protein interactions governed by a third binding partner. Plant Physiology. kiaa039.









 实验热线:4006991663
实验热线:4006991663



